2. 感染性疾病诊治协同创新中心, 浙江 杭州 310003
2. Collaborative Innovation Center for Diagnosis and Treatment of Infectious Diseases, Hangzhou 310003, China
自二十世纪四十年代抗生素作为抗感染治疗的重要手段进入临床以来,抗生素的应用范围不断扩大,近几十年来已全面推广到畜牧、水产养殖业,用于促进动物健康生长和疾病预防。在人类和动物中,抗菌药物过度使用,甚至滥用、误用,导致细菌对抗菌药物的耐药性急剧增加,已经发展成为全世界关注的公共卫生难题,人类健康受到极大威胁。抗菌药物耐药性是由抗菌药物耐药性基因(antimicrobial resistance gene, ARG)导致的,近年研究表明,ARG广泛存在于自然环境中,如土壤[1]、水[2]、沉积物[3]、动物[4]和人的肠道[5]中。尽管ARG已被证明具有悠久的历史,可以追溯到抗生素时代之前,但是抗菌药物的使用无疑加速了其在环境-动物-人体中的传播。在众多的抗菌药物类中,四环素类药物因其价格低、作用范围广、毒性低等优点,曾作为一种广谱抗生素广泛应用于临床和禽畜养殖业[6]。然而,随着四环素类抗生素的大量使用,细菌对其逐渐产生了耐药性,治疗效果大大降低,限制了四环素类抗生素在临床上的应用。目前,临床仅有少量四环素类药物用于治疗非典型肺炎、牙周感染、急性盆腔炎、痤疮等局部或全身感染[7-10]。而在兽医和禽畜养殖业中,四环素类药物仍被用作动物的抗菌促生长剂[7, 11]。自第一个耐四环素志贺痢疾杆菌(1953年)和多重耐药志贺菌属(1955年)发现以来,耐四环素菌株不断增多,并以多重耐药为主。细菌对四环素类药物耐药的机制主要是获得编码相关蛋白的四环素耐药基因,目前发现的四环素类耐药基因种类已达40多种,已知的耐药机制主要有外排泵出机制、核糖体保护机制及产生灭活四环素的钝化酶等[12]。而耐药基因与可移动成分,如可传递性质粒、转座子、接合转座子等结合,发生水平基因转移是导致四环素类耐药基因传播的重要因素之一,从而导致四环素耐药基因广泛分布于人源、动物源和环境源这些由细菌组成的微生态环境中[13]。
目前,四环素耐药基因在自然界微生态环境中的分布情况尚不明确,为深入、系统的探索四环素类耐药基因在人体、动物和环境微生态中的分布情况和耐药基因多样性,本研究拟采用宏基因组学和生物信息学分析工具,对2 036份来自于人、动物和环境微生态的宏基因组测序样本进行四环素类耐药基因研究,为临床和畜牧业感染性疾病的治疗提供理论基础。
1 材料与方法 1.1 宏基因组数据来源收集HMP[14]、META-HIT[15]、MG-RAST[16]和IMicrobe[17]四个公共数据库,以及2型糖尿病研究中[18]所测得的中国健康人肠道数据的宏基因组测序样本。HMP和META-HIT数据库主要是关注人类微生物组;MG-RAST数据库是一个开源数据库,样本来源涉及人、动物和环境;IMicrobe数据库主要关注环境样本的宏基因组研究;T2D-Chinese中的人类样本是关于2型糖尿病研究[18]中的健康对照。
1.2 四环素耐药基因来源目前已经发现44种四环素耐药基因,基因序列均下载于CARD耐药数据库[19]。
1.3 生物信息分析基于基因序列相似度比对的方法,对来自耐药基因数据库CARD的44种四环素耐药基因(tet)在人体、动物和环境中的分布进行筛查。使用自主编写的perl脚本对测序的原始数据进行过滤,然后用Bowtie2(v.2.2.6)[20]将过滤后的数据分别与44种四环素耐药基因进行比对。reads比对长度>50 bp,且匹配reads数>10条则认为该样本中存在此耐药基因,反之则认为可能是随机匹配,此样本中未检出耐药基因。
1.4 统计分析方法应用R 3.4.0软件进行数据分析,组间差异采用Fisher确切概率检验,以P≤0.05为差异有统计学意义。
2 结果 2.1 宏基因组测序样本分布情况共收集2 036份微生态样本的宏基因组测序数据,均来自公共数据库或已发表的研究项目,分别为人1 114份,动物61份和环境861份,样本多来自人类与环境,动物源样本较少。详见表 1。
| 表 1 2 036份宏基因组数据来源及类别分布情况 Table 1 Distribution of sources and categories of 2 036 metagenomic data |
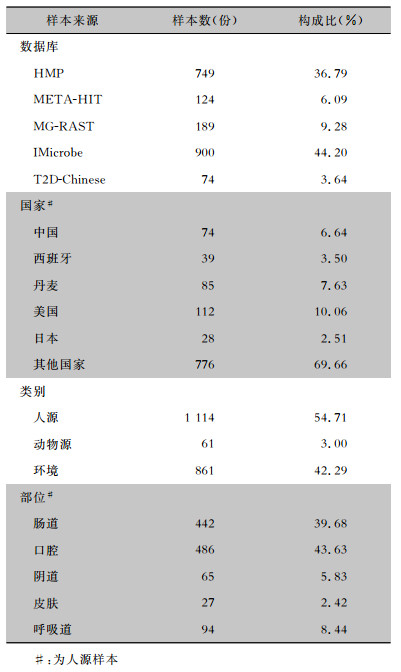
|
2 036份样本中,910份样本检出四环素耐药基因,检出率44.70%。共检出28种四环素耐药基因,248份样本至少检出10种以上四环素耐药基因。人源样本中检出率最高,达78.99%(880/1 114),且耐药基因分布最丰富,28种四环素类耐药基因全部检出,占比较大;其次是动物样本,检出率40.98%(25/61),检出6种四环素类耐药基因;环境样本中检出率最低,861份样本中仅5份(0.58%)检出,且耐药基因多样性的分布比较窄,仅匹配到2种四环素类耐药基因,分别为tetG(4份)、tetK(1份)。见图 1。
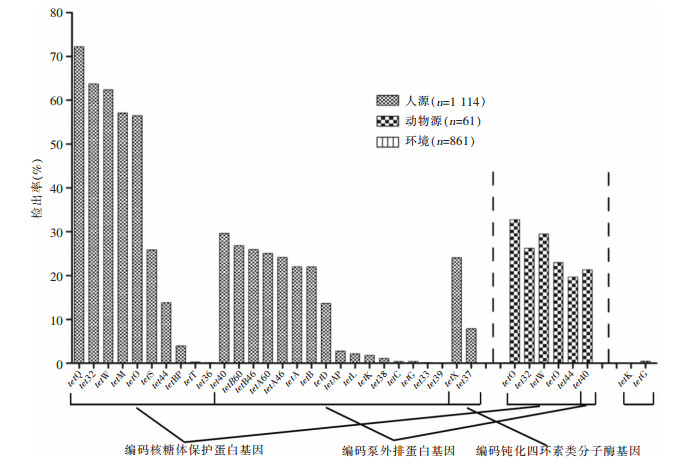 |
| 图 1 不同来源样本中四环素耐药基因和耐药机制的分布 Figure 1 Distribution of tetracycline resistance genes and resistance mechanisms in specimens from different sources |
28种四环素类耐药基因根据耐药机制进行分类,结果显示,10种为编码核糖体保护蛋白基因(tetM、tetO、tetQ、tetS、tetT、tetW、tetBP、tet32、tet36、tet44),16种为编码泵外排蛋白基因(tetA、tetB、tetC、tetD、tetG、tetK、tetL、tetAP、tetA46、tetA60、tetB46、tetB60、tet33、tet38、tet39、tet40),以及2种编码钝化四环素类分子酶基因(tetX、tet37)。编码核糖体保护蛋白基因耐药机制占据绝对优势,10种基因中有5种(tetQ、tet32、tetW、tetO、tetM)在50%以上的人类样本中检出,其中4种在动物中也有较高的检出率(16%以上);而编码泵外排蛋白基因分布较广,基因型别最丰富,在人源样本中广泛分布,7种耐药基因占比20%以上,动物源样本中仅检出1种耐药基因(tet40),同时此耐药机制也是环境源样本仅检出的1种;编码钝化四环素类分子酶基因型别较少,且只匹配到了人类样本上。见图 1。
2.4 不同国别四环素耐药基因的分布1 114份人源样本中有776份样本国别信息不完整,剔除此部分数据(如HMP和IMicrobe中缺乏清楚的国别信息的样本),338份人源样本具有国别信息,其中,中国74份,西班牙39份,丹麦85份,美国112份,日本28份。分析结果显示,不同国家的微生态样本耐药基因分布存在很大的差异。中国来源的微生态样本共匹配上24种四环素耐药基因型,有5种耐药基因型别在样本中所占百分比为100%,即在所有中国样本中均检出了该耐药基因,分别是tet32、tet40、tetO、tetQ和tetW;其次是丹麦,匹配上17种四环素耐药基因型,tet32、tetO、tetQ和tetW四种基因型占比100%;西班牙共匹配上16种四环素耐药基因型,tet32、tetO和tetW三种基因型占比100%;美国和日本耐药基因型别最少,分别匹配上5种和3种四环素耐药基因型,且耐药率很低,基本在20%以下。同时研究发现,tetO、tetQ和tetW三种基因在5个国家中均有检出,而tetL、tetS、tet33、tet36、tetA60、tetT、tet38为中国特有四环素耐药基因。详见图 2。
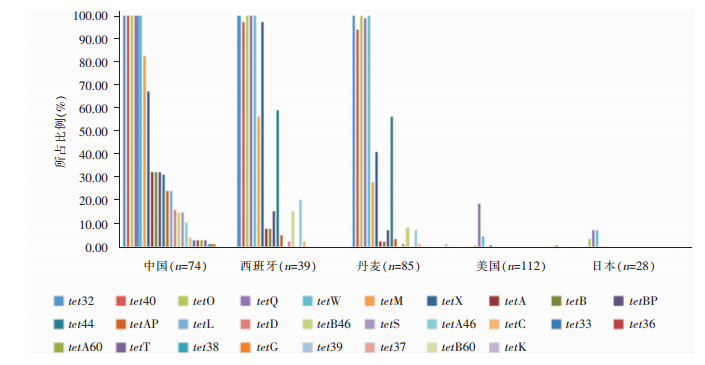 |
| 图 2 不同国别四环素耐药基因分布情况 Figure 2 Distribution of tetracycline resistance genes in different countries |
1 114份人源样本根据采样部位进行分类,分为肠道、口腔、阴道、皮肤和呼吸道5大类。人体不同部位的四环素耐药基因分布是不均匀的,肠道中的耐药基因型分布最丰富,共有26种四环素耐药基因匹配到肠道样本中,其中有5种耐药基因型所占百分比>70%,即tetQ、tetW、tetO、tet32和tet40;其次是口腔,共检出21种四环素耐药基因,tetM、tetQ和tet32三种耐药基因型占比>70%;阴道和皮肤中均检出10种耐药基因,呼吸道中检出9种耐药基因,阴道、皮肤及呼吸道检出的耐药基因所占百分比均低于45%。见图 3。
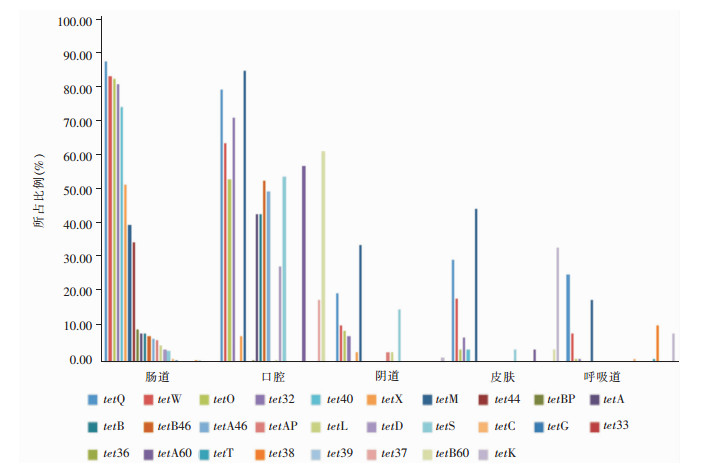 |
| 图 3 不同部位四环素耐药基因分布情况 Figure 3 Distribution of tetracycline resistance genes in different sites |
抗菌药物的使用和细菌耐药性是相互依存、相互制约的两个方面。人类、动物和环境之间也不是孤立存在的,抗菌药物耐药基因可以通过水平转移在细菌间传播,也可以通过食物和接触的方式从动物微生态环境到人群微生态环境传播,而生活污水、废水,以及排泄物又造成耐药基因从动物和人类的微生态环境向环境中进一步传播,环境微生态中的抗菌药物积累到一定浓度也会通过生物链对人体和其他生物体构成潜在危害。采用宏基因组学分析方法,对来源于人、动物、环境的2 036份微生态样本的宏基因组测序数据进行四环素类耐药基因筛查和耐药机制研究,结果显示,不同来源的微生态样本中四环素耐药基因分布不同,人类样本中的耐药基因分布最丰富,其次是动物样本和环境样本。人类样本中的四环素耐药基因最丰富,且多为多种耐药基因并存,可能与长期以来人类抗菌药物滥用和误用有关。同时四环素类药物作为动物的抗菌促生长剂,广泛用于禽畜养殖业,因此,在动物样本中检测到了高比例的四环素耐药基因。相比于人类和动物样本,环境样本中耐药基因占比很小,推测可能是由于环境样本主要来源于海洋、大气、土壤等,并未暴露在抗菌药物聚集的地方;其次也可能受实验和测序深度的影响,造成检出率比较低。目前,已发现44种四环素耐药基因,本研究检出28种耐药基因,以编码核糖体保护蛋白的耐药机制占据主导,其次为编码泵外排蛋白耐药,且此机制耐药基因检出型别最多,说明四环素耐药性的产生主要是由于获得了此两种耐药机制编码的四环素耐药基因而介导。
同时研究发现,不同国家间四环素耐药基因的分布也是不均衡的,中国、丹麦和西班牙的微生态样本中都检出不同类型的四环素耐药基因,tetL、tetS、tet33、tet36、tetA60、tetT、tet38为仅在中国微生态样本中检出的耐药基因,美国和日本耐药比例相对比较低,与胡永飞等[21]研究结果一致,可能是国家的医疗制度和饮食习惯不同造成的。tetO、tetQ和tetW 3种基因在5个国家中均有检出,说明此3种基因是四环素耐药基因中最普遍,分布最广的基因型。人体不同部位耐药基因分布研究显示,肠道和口腔微生态中的耐药基因都很丰富,肠道中的耐药基因型分布最普遍的是tetQ、tetW、tetO、tet32和tet40,而口腔中普遍存在的是tetM、tetQ和tet32 3种耐药基因型。耐药基因型差异较大,可能是人体不同部位的微生物菌群结构的差异造成的。此结果与Seville等[22]在2009年的研究结果一致,研究分析欧洲六国健康人群的粪便和唾液样品中的四环素耐药基因,发现口腔中最普遍存在的是tetM,肠道中普遍存在的依次是tetW、tetQ和tetO。人体肠道中定植着大量的微生物群体,这些正常菌群作为耐药基因的储存库,不断的将耐药基因水平转移给致病菌,通过与外界环境的交互作用,在人和动物中交叉传播,尤其是释放到环境中的耐药菌危害更严重,可造成耐药基因的迅速转移散播,因此,肠道菌群中分布最丰富,占比最高的耐药基因应受到足够的重视。
研究四环素类耐药基因在人体、动物和环境微生态中的分布情况,以及耐药基因在不同来源、不同国别、不同人体部位的分布及耐药机制,旨在为临床和畜牧业等感染性疾病的治疗提供一定的理论基础。细菌耐药性是全世界共同关注的议题,促进临床合理合规使用抗菌药物,以及减少动物例行使用抗菌药物,是减缓人类和动物中抗药性产生速度的重大措施。同时也呼吁相关部门结合环境中抗菌药物的污染现状,建立抗菌药物环境风险评估机制,对抗菌药物耐药进行有效的监控,提出符合我国国情的控制策略和预防措施。
| [1] |
Riesenfeld CS, Goodman RM, Handelsman J. Uncultured soil bacteria are a reservoir of new antibiotic resistance genes[J]. Environ Microbiol, 2004, 6(9): 981-989. DOI:10.1111/j.1462-2920.2004.00664.x |
| [2] |
Baquero F, Martinez J, Cantón R. Antibiotics and antibiotic resistance in water environments[J]. Curr Opin Biotechnol, 2008, 19(3): 260-265. DOI:10.1016/j.copbio.2008.05.006 |
| [3] |
Pei R, Kim S, Carlson K, et al. Effect of river landscape on the sediment concentrations of antibiotics and corresponding antibiotic resistance genes (ARG)[J]. Water Res, 2006, 40(12): 2427-2435. DOI:10.1016/j.watres.2006.04.017 |
| [4] |
Caprioli A, Busani L, Martel JL, et al. Monitoring of antibio-tic resistance in bacteria of animal origin:epidemiological and microbiological methodologies[J]. Int J Antimicrob Agents, 2000, 14(4): 295-301. DOI:10.1016/S0924-8579(00)00140-0 |
| [5] |
Sommer MOA, Dantas G, Church GM. Functional characteri-zation of the antibiotic resistance reservoir in the human microflora[J]. Science, 2009, 325(5944): 1128-1131. DOI:10.1126/science.1176950 |
| [6] |
代敏, 王雄清, 殷桂兰. 四环素耐药基因的生化和遗传机制研究进展[J]. 绵阳师范学院学报, 2006, 25(5): 72-78. DOI:10.3969/j.issn.1672-612X.2006.05.021 |
| [7] |
Roberts MC. Acquired tetracycline and/or macrolide-lincosamides-streptogramin resistance in anaerobes[J]. Anaerobe, 2003, 9(2): 63-69. DOI:10.1016/S1075-9964(03)00058-1 |
| [8] |
肖忠革, 金剑, 陈臻. 四环素类药物治疗牙周炎的作用机制及临床应用进展[J]. 现代中西医结合杂志, 2005, 14(3): 409-410, 420. DOI:10.3969/j.issn.1008-8849.2005.03.110 |
| [9] |
徐霞. 甲硝唑联合四环素治疗急性盆腔炎的疗效[J]. 实用临床医学, 2015, 16(8): 68-69. |
| [10] |
张辰蔚, 李洋. 四环素及其衍生物在类风湿关节炎的应用[J]. 中华临床免疫和变态反应杂志, 2016, 10(3): 286-291. |
| [11] |
韦嫔. 四环素类抗生素在养猪业的应用[J]. 饲料与畜牧·规模养猪, 2015(7): 55-58. |
| [12] |
Chopra I, Roberts M. Tetracycline antibiotics:mode of action, applications, molecular biology, and epidemiology of bacterial resistance[J]. Microbiol Mol Biol Rev, 2001, 65(2): 232-260. DOI:10.1128/MMBR.65.2.232-260.2001 |
| [13] |
Thomas CM, Nielsen KM. Mechanisms of, and barriers to, horizontal gene transfer between bacteria[J]. Nat Rev Microbiol, 2005, 3(9): 711-721. DOI:10.1038/nrmicro1234 |
| [14] |
Human Microbiome Project Consortium. A framework for human microbiome research[J]. Nature, 2012, 486(7402): 215-221. DOI:10.1038/nature11209 |
| [15] |
Ehrlich SD. MetaHIT: The European Union project on meta-genomics of the human intestinal tract[M]//Nelson KE, Metagenomics of the Human Body, Springer, New York, NY, 2011: 307-316.
|
| [16] |
Keegan KP, Glass EM, Meyer F. MG-RAST, a metageno-mics service for analysis of microbial community structure and function[J]. Methods Mol Biol, 2016, 1399: 207-233. |
| [17] |
Hurwitz B. iMicrobe: Advancing clinical and environmental microbial research using the iPlant cyberinfrastructure[C]. International Plant & Animal Genome Conference XXⅡ, 2014.
|
| [18] |
Qin J, Li Y, Cai Z, et al. A metagenome-wide association study of gut microbiota in type 2 diabetes[J]. Nature, 2012, 490(7418): 55-60. DOI:10.1038/nature11450 |
| [19] |
Jia B, Raphenya AR, Alcock B, et al. CARD 2017:expansion and model-centric curation of the comprehensive antibiotic resistance database[J]. Nucleic Acids Res, 2017, 45(D1): D566-D573. DOI:10.1093/nar/gkw1004 |
| [20] |
Langmead B, Salzberg SL. Fast gapped-read alignment with Bowtie 2[J]. Nat Methods, 2012, 9(4): 357-359. DOI:10.1038/nmeth.1923 |
| [21] |
Hu Y, Yang X, Qin J, et al. Metagenome-wide analysis of antibiotic resistance genes in a large cohort of human gut micro-biota[J]. Nat Commun, 2013, 4: 2151. DOI:10.1038/ncomms3151 |
| [22] |
Seville L, Patterson AJ, Scott KP, et al. Distribution of tetracycline and erythromycin resistance genes among human oral and fecal metagenomic DNA[J]. Microb Drug Resist, 2009, 15(3): 159-166. DOI:10.1089/mdr.2009.0916 |



