Whole genome analysis of a Coxsackievirus A4 strain from Yunnan
-
摘要:
目的 了解中国云南2022年分离到的一株柯萨奇病毒A组4型(Coxsackievirus A4, CVA4)病毒株的全基因组序列特征,探讨CVA4系统发育特征。 方法 对CVA4分离株194R3/YN/CHN/2022进行全基因组序列扩增测序,并应用Mega 7.0、Geneious 9.1.4及Simplot 3.5.1等软件拼接比对,构建CVA4分离株系统进化树,分析其全基因组序列特征。 结果 194R3/YN/CHN/2022分离株为CVA4,属于C2基因亚型,与我国近年的优势基因亚型一致。重组分析显示,在P2、P3非结构编码区,CVA4病毒分离株可能与EVA114原型株V13-0285、CVA16原型株G-10、CVA14原型株G-14发生过重组。 结论 云南分离的194R3/YN/CHN/2022属于CVA4中国流行的C2基因亚型,但发生了一定变异。 -
关键词:
- 柯萨奇病毒A组4型 /
- 全基因组序列 /
- 系统进化分析 /
- 194R3/YN/CHN/2022
Abstract:Objective To understand the whole genome sequence characteristics of a Coxsackievirus A4 (CVA4) isolated from Yunnan, China in 2022, and explore the phylogenetic characteristics of CVA4. Methods The whole genome sequence of CVA4 isolate 194R3/YN/CHN/2022 was amplified and sequenced, and the phylogenetic tree of CVA4 isolate was constructed by using Mega 7.0, Geneious 9.1.4 and Simplot 3.5.1 softwares. The whole genome sequence characteristics were analyzed. Results The 194R3/YN/CHN/2022 isolate was identified as CVA4, belonging to the C2 gene subtype, which was consistent with the dominant gene subtype in recent years in China. Recombination analysis showed that recombination of CVA4 virus isolate with EVA114 prototype (V13-0285), CVA16 prototype (G-10), and CVA14 prototype (G-14) at the non-structural coding regions of P2 and P3 may have occurred. Conclusion The 194R3/YN/CHN/2022 isolated from Yunnan belongs to the C2 gene subtype, which is the prevalent CVA4 in China, but with certain mutations. -
Keywords:
- Coxsackievirus A4 /
- whole genome sequence /
- phylogenetic analysis /
- 194R3/YN/CHN/2022
-
手足口病(hand, foot, and mouth disease, HFMD)是全球广泛流行的儿童传染病,多发于5岁以下儿童,主要临床表现为发热、手足和口腔等部位出现疱疹或皮疹[1-3],而肠道病毒(Enterovirus,EV)是其病原体。其中柯萨奇病毒A组4型(Coxsackievirus A4, CVA4)除可引起HFMD,也与疱疹性咽峡炎、急性局部皮疹、急性弛缓性麻痹、无菌性脑膜炎、脑炎等疾病相关[4]。近年来,流行病学研究[5-7]显示,CVA4在HFMD中的检出率逐渐升高,CVA4引起的HFMD在多个国家及地区流行和暴发。
因此,本研究分析了2022年云南省一株CVA4病毒分离株的全基因组序列,探索CVA4流行株的进化特点及基因变异趋势,为云南省CVA4流行株的遗传变异情况提供实验室数据,对CVA4的疫苗研发和相关疾病的防控具有参考意义。
1. 材料与方法
1.1 病毒株及细胞
病毒分离株来源于2022年云南省HFMD监测中一例2岁10个月男性儿童HFMD轻症患者,其临床特点为皮疹,见于手掌、足趾,伴有发热。人横纹肌肉瘤(human rhabdomyosarcoma, RD)细胞由本实验室保存、提供。
1.2 主要仪器与设备
二氧化碳恒温细胞培养箱购自美国Thermo公司,PCR仪购自美国Bio-Rad公司,生物安全柜购自苏州安泰空气技术有限公司。
1.3 病毒增殖与病毒RNA提取
病毒分离株经RD细胞增殖培养,收获阳性病毒培养液,取200 μL,按照Axygen Body Fluid viral DNA/RNA Miniprep Kit(Axygen, 美国)说明书,提取病毒RNA。
1.4 序列扩增与血清型鉴定
根据CVA4分离株JN19075/Shandong/China/2019(基因收录号No. 730874.1)与参考文献[8]设计针对该病毒分离株全基因组各片段的引物,分段扩增其全基因组序列并测序。扩增及测序引物序列见表 1。
表 1 病毒分离株全基因组序列扩增和测序引物Table 1 Primers for whole genome amplification and sequencing of virus strains引物名称 序列(5’→3’) 核苷酸位点 引物方向 A-OAS#* GGRTANCCRTCRTARAACCAYTG 2 268~2 287 下游 A-OS#* CCNTGGATHAGYAACCANCAYT 3 109~3 089 上游 E201F#* TTAAAACAGCCTGTGGGTTG 1~20 上游 A4N2R#* GGGTTATCTCAGGTGTGTCT 2 407~2 388 上游 A4N1r* GTGATAGCGGAATGAATC 683~666 下游 A4N1f* CACTAATATTAATTATTA 815~832 上游 A4N1ff* CACAGCGATCTGGGTCTC 749~776 上游 A4N1f2* ACCATTGAGCCAGGCATT 1 457~1 474 上游 A4N3F#* CCCTAAACCTGACGCCCGAGAATC 2 907~2 930 上游 E208R#* ACCGAATGCGGAGAATTTAC 7 384~7 365 下游 A4N3f* GAGGGATCTCCTCGTGTC 3 455~3 472 上游 A4N4f* GCCACTACTGGAGAATCA 4 250~4 267 上游 A4N5f* TGCGGGAAAGCTATTCAG 4 947~4 964 上游 A4N6f* GTTGGGAAGGTCATTGGC 5 862~58 49 上游 A4N7f* GTCCAGTTTGGTTCAGGG 6 689~6 706 上游 CA168R#* GGTTATAACAAATTTACCCCC 7 413~7 393 下游 注:#表示扩增引物;*表示测序引物。 使用RT-PCR扩增试剂盒(PrimeScriptTM One step RT-PCR Kit Ver.2)扩增全基因组序列,扩增程序为:55℃ 30 min,94℃ 4 min,94℃ 30 s,50℃ 30 s,72℃ 1 min,循环30次,最后72℃ 10 min进行终末延伸。由昆明擎科生物科技公司完成测序。使用NCBI在线网站(https://www.ncbi.nlm.nih.gov/)BLAST工具鉴定病毒分离株血清型。
1.5 全基因组序列测定与分析
应用DNAStar 7.1软件对分离株全基因组进行拼接及组装。从GenBank数据库中随机选取45株CVA4毒株作为参考株,信息见表 2。应用Geneious 9.1.4软件对本研究病毒分离株序列及参考株序列进行VP1区序列比对及同源性分析。应用Mega 7.0软件,采用最大似然法(maximum likelihood, ML)构建进化树,建树自展值(Bootstrap)检验1 000次,对VP1序列进行系统进化分析。
表 2 VP1系统进化分析参考株信息Table 2 Information of reference strains used in phylogenetic analysis based on VP1毒株名称 国家 采样年份 基因登录号 毒株名称 国家 采样年份 基因登录号 High Point 美国 1948 AY421762 701/SH/CHN/2010 中国 2010 KJ541163 N-48 印度 2007—2009 JN203505 SaX10-8 中国 2010 KJ818325 N-56 印度 2007—2009 JN203507 4208/CVA4/SH/CHN/2012 中国 2012 KP398837 N-253 印度 2007—2009 JN203509 13-11-HeN-2013 中国 2013 MK388451 N-980 印度 2007—2009 JN203513 13219/SD/CHN/2013 中国 2013 KY978560 C082/CHW/AUS/2016 澳大利亚 2016 MH111027 CV-A4/P1033/2013/China 中国 2013 KP289442 JR 日本 2008 AB457644 13-66-CQ-2013 中国 2013 MK388454 11023.1 肯尼亚 1999 GQ176232 14-41-JX-2014 中国 2014 MK391071 SPb-5171/14mi/UZ-7/14/RU 俄罗斯 2015 KR185979 CVA4/FT/CHN/05 中国 2015 KP676984 SPb 5161/13EVI/Mur-26/14/RU 俄罗斯 2013 KR185978 CVA4/FT/CHN/07 中国 2015 KP676985 USA/TN/2015-OB2038 美国 2015 KY271949 15-116-LN-2015 中国 2015 MK391072 98401/SD/CHN/1998/CA4 中国 1998 GQ253377 CVA4/FT/CHN/27 中国 2015 KP676986 GD10-114 中国 1998—2012 KJ818310 16-69-TJ-2016 中国 2016 MK388484 03188/SD/CHN/2003 中国 2003 KY978541 AA 中国 2016 MG550920 JL-CHN-AFP-CVA4-3/2006 中国 2006 JQ715708 CV-A4/2018/herpangina/17 中国 2018 MN964086 JL-CHN-AFP-CVA4-1/2006 中国 2006 JQ715709 SF047 中国 2018 LC361273 SC06-44 中国 2006 LC175758 180-QJ-YN-CHN-2019-CV-A4 中国 2019 LC603077 SC07-282 中国 2007 LC175762 56/QJ/YN/CHN/2020/CV-A4 中国 2020 LC626199 JL-CHN-H-CVA4-1/2007 中国 2007 JQ715710 180/QJ/YN/CHN/2020/CV-A4 中国 2020 LC626238 1-E9-CA4 中国 2008 KT353722 69-QJ-YN-CHN-2021-CVA4 中国 2021 LC707407 F1212/SD/CHN/2008 中国 2008 KY978537 189-QJ-YN-CHN-2021-CVA4 中国 2021 LC707448 JB141230145 中国 2008—2012 KC867064 23-QJ-YN-CHN-2021-CVA4 中国 2021 LC707391 CVA4/SZ/CHN/09 中国 2009 HQ728260 应用相同软件和参数对本研究分离株和GenBank数据库中获得的CVA4分离株及所有EV-A原型株的全长P1、P2、P3区域进行系统进化分析和发育分析。应用Simplot 3.5.1软件进行重组分析验证。
2. 结果
2.1 血清型鉴定
RD细胞分离株VP1序列经NCBI在线网站确定其血清型为CVA4,命名为194R3/YN/CHN/2022。
2.2 CVA4病毒分离株194R3/YN/CHN/2022全基因组序列分析
该分离株基因含7 434个核苷酸,编码2 478个氨基酸,5’UTR长度为747个核苷酸,一个开放阅读框(open reading frame, ORF) 长度为6 602个核苷酸,3’UTR长度为85个核苷酸。
不同CVA4病毒分离株全基因组序列比对结果显示,194R3/YN/CHN/2022分离株与山东分离株JN20186/Shandong/China/2020相似性最高;194R3/YN/CHN/2022分离株与CVA4原型株High Point核苷酸、氨基酸同源性分别为84.30%、97.2%;194R3/YN/CHN/2022各基因区域与其他CVA4分离株核苷酸相似性为97.60%~99.52%。194R3/YN/CHN/2022分离株与我国其他CVA4病毒分离株的全基因序列对比结果见表 3。
表 3 194R3/YN/CHN/2022分离株与其他CVA4病毒分离株不同基因组区域的同源性比较Table 3 Homology comparison of different genomic regions between 194R3/YN/CHN/2022 and other CVA4 strains基因区域 血清型 毒株 核苷酸同源性(%) 基因登录号 5’UTR CVA4 CV-A4/2018/herpangina/25 98.39 MN964081.1 VP4 CVA4 S270/Changsha/CHN/2019 99.52 MT920660.1 VP2 CVA4 CV-A4/2018/herpangina/16 99.48 MN964082.1 VP3 CVA4 JN20186/Shandong/China/2020 98.89 ON730850.1 VP1 CVA4 SJ2021-41/SH/CHN/CVA4/2021 97.92 MT591575.1 2A CVA4 E140/YN/CHN/2019 97.60 MT591575.1 2B CVA4 CV-A4/2018/herpangina/16 98.32 MN964082.1 2C CVA4 JN20186/Shandong/China/2020 98.48 ON730850.1 3A CVA4 JN19075/Shandong/China/2019 98.45 ON730874.1 3B CVA4 CV-A4/2018/herpangina/16 98.48 MN964082.1 3C CVA4 JN19737/Shandong/China/2019 98.18 ON730851.1 3D CVA4 JN19737/Shandong/China/2019 98.56 ON730851.1 3’UTR CVA4 CV-A4/2018/herpangina/25 98.81 MN964081.1 P1 CVA4 JN20186/Shandong/China/2020 98.28 ON730850.1 P2 CVA4 JN20186/Shandong/China/2020 97.81 ON730850.1 P3 CVA4 JN19737/Shandong/China/2019 98.41 ON730851.1 基因组 CVA4 JN20186/Shandong/China/2020 98.20 ON730850.1 2.3 系统进化分析
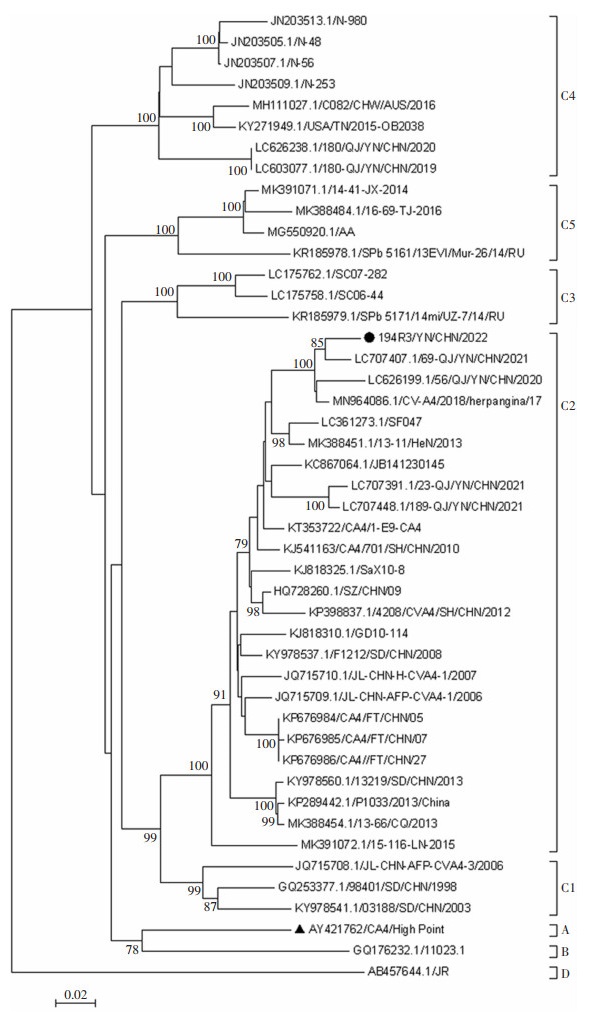
应用软件Mega 7.0将194R3/YN/CHN/2022分离株和45株参考株的全长VP1序列(915 bp)构建系统发育树,见图 1。所有CVA4病毒分离株分为A、B、C、D 4个基因型,其中,1948年分离自美国北卡罗来纳州[9]的CVA4原型株High Point单独构成A基因型;肯尼亚分离株11023.1单独构成B基因型。C基因型的组成较复杂,可进一步分成C1、C2、C3、C4、C5 5个基因亚型。C1基因亚型由一株2006年吉林省CVA4分离株与山东省1998、2003年的两株CVA4分离株组成;C3基因亚型由一株2014年俄罗斯CVA4分离株与两株四川省于2006、2007年分离的CVA4分离株组成;C4基因亚型由2007—2009年期间印度CVA4分离株,2015、2016年中国台湾CVA4分离株,以及2019、2020年云南CVA4分离株组成;C5基因亚型由一株2017年河南省CVA4分离株、2016年一株天津CVA4分离株、2014年一株江西省CVA4分离株,以及2015年一株俄罗斯CVA4分离株组成;其余CVA4分离株为C2基因亚型。当前世界范围的优势基因型是C基因型,我国近几年的优势基因亚型为C2基因亚型,这与之前的研究[10]结果相同。194R3/YN/CHN/2022分离株属于C2基因亚型。
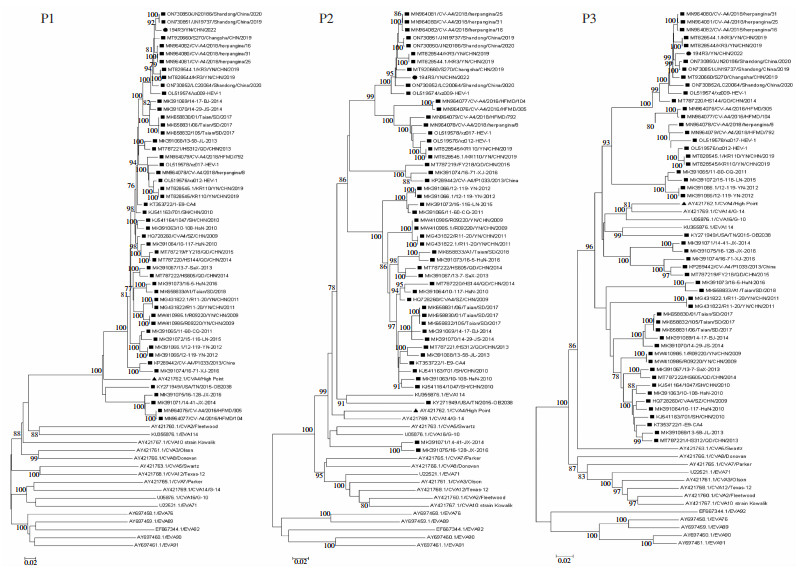
对本研究分离到的CVA4病毒分离株194R3/YN/CHN/2022及根据文献报道[11]的GenBank数据库中下载的所有EV-A原型株和随机选取的CVA4型病毒代表株的P1、P2、P3区,应用软件Mega 7.0构建系统进化树,见图 2。系统进化树显示,在P1结构编码区,GenBank数据库中随机选取的CVA4型病毒代表株与194R3/YN/CHN/2022分离株与CVA4原型株High Point都聚为一簇,而其他的EV-A原型株不与CVA4分离株聚类,与VP1分型结果一致;在P2非结构编码区,194R3/YN/CHN/2022分离株与CVA4原型株High Point的亲缘关系较远,与肠道病毒A114型(Enterovirus 114, EVA114)原型株V13-0285及GenBank数据库中随机选取的CVA4型病毒代表株聚类在一起,提示CVA4分离株P2区段可能发生过不同型别间的重组;而在P3非结构编码区,本研究所分离的CVA4病毒分离株194R3/YN/CHN/2022与GenBank数据库中随机选取的2019年及之后的CVA4型病毒代表株亲缘关系较近,还与EVA114原型株V13-0285、CVA16原型株G-10、CVA14原型株G-14聚类在同一进化分支上,而未与CVA4原型株High Point聚类在同一进化分支,但相比于GenBank数据库中随机选取的2018年及之前的CVA4病毒代表株,194R3/YN/CHN/2022与CVA4原型株High Point亲缘关系更近。这些结果表明CVA4流行株可能在非结构编码区与EV-A原型株之间发生过重组。
2.4 重组分析
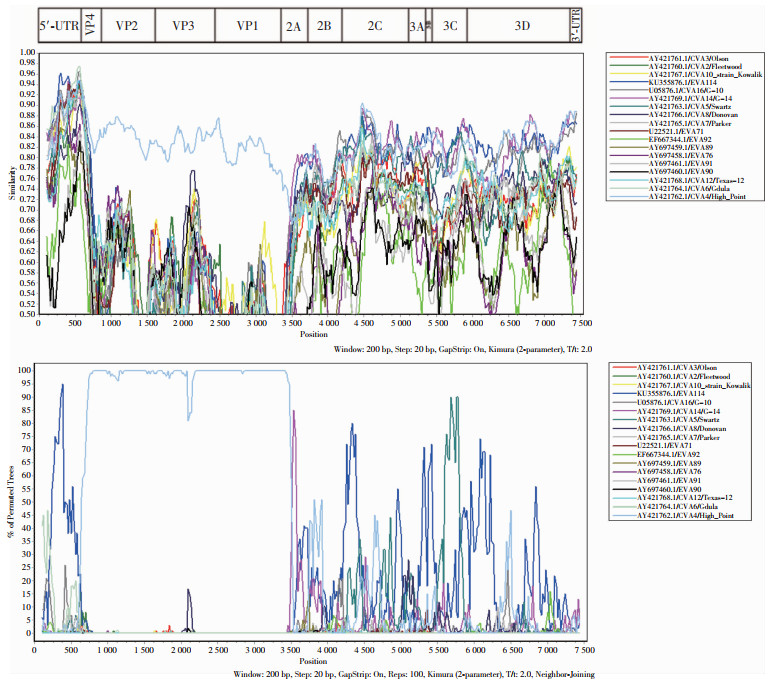
为进一步探究194R3/YN/CHN/2022分离株在非结构编码区与EV-A原型株之间是否可能发生重组,以CVA4 194R3/YN/CHN/2022分离株为质询序列,与所有EV-A原型株的全基因组序列进行Simplot和Bootscan分析,见图 3。结果显示,在P1结构编码区,194R3/YN/CHN/2022分离株与CVA4原型株High Point的相似性最高,而在P2、P3非结构编码区,194R3/YN/CHN/2022分离株与其他多个EV-A原型株存在较高的相似性。在2C与3D区段与EVA114原型株的相似性最高,通过相似性分析进一步验证了该结果,提示本研究所分离得到的194R3/YN/CHN/2022分离株在进化过程中可能与其他EV-A病毒分离株发生了重组,其中最有可能在2C与3D区段与EVA114原型株发生重组,且重组位点位于P2、P3非结构编码区。
3. 讨论
EV71疫苗获批上市后,HFMD病原谱逐渐发生变化[12]。在中国大陆多地都有CVA4病毒引起HFMD流行的报道[13-16]。2018—2021年昆明市HFMD监测中发现CVA4引发的HFMD已成为其他EV(非EV-71、CVA16)引发HFMD的重要组成[17]。因此,加强对CVA4的病原学监测及分子流行病学研究,有助于预防和控制CVA4引起的HFMD。
EV的VP1区域是CVA4主要抗原决定簇所在区域,根据VP1序列对EV进行血清型鉴定和基因分型应用广泛[18]。本研究基于CVA4完整VP1序列,将CVA4划分为A~D 4个基因型,并发现中国近几年的优势基因亚型为C2亚型,且194R3/YN/CHN/2022分离株也属于C2亚型,与此前研究[13]结果一致。近年来,GenBank数据库中上传的CVA4分离株大部分为C2基因亚型,提示C2基因亚型有一定进化优势,并促进其传播和流行。但是也发现不同区域或年份分离株的核苷酸和氨基酸序列存在差异,提示CVA4病毒株已经发生一定进化。
基因重组是EV进化的主要方式。病毒复制过程中趋向于更好地适应环境的选择性优势,导致出现新的流行毒株,甚至引起相关疾病的暴发流行[19-21]。基于194R3/YN/CHN/2022分离株与GenBank数据库中随机选取的CVA4病毒分离株的P1、P2、P3区构建的系统进化树显示,在P1编码区,194R3/YN/CHN/2022分离株与之前分离获得的CVA4分离株之间无较大差异;而在P2、P3非结构编码区,194R3/YN/CHN/2022分离株与其他多个EV-A原型株之间存在较高的相似性。相似性分析显示,194R3/YN/CHN/2022分离株可能与EVA114原型株在2C和3D非结构蛋白编码区发生了重组事件。这与青岛分离的病毒株可能在2C和3D区发生重组的研究[22]结果一致;2020—2021年上海市EV监测中,也发现C2基因亚型CVA4病毒在非结构编码区发生了重组[23]。这些结果提示,CVA4分离株的C2基因亚型流行优势可能来源于其不断积累的基因重组导致的遗传变异,使其得以生存,且具有较广泛的传播能力,但是否会形成稳定的进化趋势还需更多监测数据进一步研究。
此外,CVA4分离株感染5岁以下儿童,与EV总体感染特征一致,可引起患者HFMD,轻症出现皮疹伴随发热,重症可导致心肌炎、胰腺炎、肝功能损伤及弛缓性麻痹[24-25]。194R3/YN/CHN/2022的致病性符合CVA4分离株引起的轻症HFMD症状,出现皮疹,见于手掌、足趾,伴有发热。
本研究针对2022年从云南HFMD监测中得到的CVA4毒株194R3/YN/CHN/2022,测定其全基因组序列,并分析其全基因组序列特征、系统进化情况及可能的重组事件,更新了我国云南地区的CVA4监测数据,为CVA4分子流行病学研究和疾病防控工作提供了实验室数据。
致谢:冯昌增和楚昭阳参与了试验。冯昌增、刘煜菡、张名、李丽、马绍辉协助解释研究结果,并协助完成写作。所有作者都审核并通过稿件的最终版本。 -
表 1 病毒分离株全基因组序列扩增和测序引物
Table 1 Primers for whole genome amplification and sequencing of virus strains
引物名称 序列(5’→3’) 核苷酸位点 引物方向 A-OAS#* GGRTANCCRTCRTARAACCAYTG 2 268~2 287 下游 A-OS#* CCNTGGATHAGYAACCANCAYT 3 109~3 089 上游 E201F#* TTAAAACAGCCTGTGGGTTG 1~20 上游 A4N2R#* GGGTTATCTCAGGTGTGTCT 2 407~2 388 上游 A4N1r* GTGATAGCGGAATGAATC 683~666 下游 A4N1f* CACTAATATTAATTATTA 815~832 上游 A4N1ff* CACAGCGATCTGGGTCTC 749~776 上游 A4N1f2* ACCATTGAGCCAGGCATT 1 457~1 474 上游 A4N3F#* CCCTAAACCTGACGCCCGAGAATC 2 907~2 930 上游 E208R#* ACCGAATGCGGAGAATTTAC 7 384~7 365 下游 A4N3f* GAGGGATCTCCTCGTGTC 3 455~3 472 上游 A4N4f* GCCACTACTGGAGAATCA 4 250~4 267 上游 A4N5f* TGCGGGAAAGCTATTCAG 4 947~4 964 上游 A4N6f* GTTGGGAAGGTCATTGGC 5 862~58 49 上游 A4N7f* GTCCAGTTTGGTTCAGGG 6 689~6 706 上游 CA168R#* GGTTATAACAAATTTACCCCC 7 413~7 393 下游 注:#表示扩增引物;*表示测序引物。 表 2 VP1系统进化分析参考株信息
Table 2 Information of reference strains used in phylogenetic analysis based on VP1
毒株名称 国家 采样年份 基因登录号 毒株名称 国家 采样年份 基因登录号 High Point 美国 1948 AY421762 701/SH/CHN/2010 中国 2010 KJ541163 N-48 印度 2007—2009 JN203505 SaX10-8 中国 2010 KJ818325 N-56 印度 2007—2009 JN203507 4208/CVA4/SH/CHN/2012 中国 2012 KP398837 N-253 印度 2007—2009 JN203509 13-11-HeN-2013 中国 2013 MK388451 N-980 印度 2007—2009 JN203513 13219/SD/CHN/2013 中国 2013 KY978560 C082/CHW/AUS/2016 澳大利亚 2016 MH111027 CV-A4/P1033/2013/China 中国 2013 KP289442 JR 日本 2008 AB457644 13-66-CQ-2013 中国 2013 MK388454 11023.1 肯尼亚 1999 GQ176232 14-41-JX-2014 中国 2014 MK391071 SPb-5171/14mi/UZ-7/14/RU 俄罗斯 2015 KR185979 CVA4/FT/CHN/05 中国 2015 KP676984 SPb 5161/13EVI/Mur-26/14/RU 俄罗斯 2013 KR185978 CVA4/FT/CHN/07 中国 2015 KP676985 USA/TN/2015-OB2038 美国 2015 KY271949 15-116-LN-2015 中国 2015 MK391072 98401/SD/CHN/1998/CA4 中国 1998 GQ253377 CVA4/FT/CHN/27 中国 2015 KP676986 GD10-114 中国 1998—2012 KJ818310 16-69-TJ-2016 中国 2016 MK388484 03188/SD/CHN/2003 中国 2003 KY978541 AA 中国 2016 MG550920 JL-CHN-AFP-CVA4-3/2006 中国 2006 JQ715708 CV-A4/2018/herpangina/17 中国 2018 MN964086 JL-CHN-AFP-CVA4-1/2006 中国 2006 JQ715709 SF047 中国 2018 LC361273 SC06-44 中国 2006 LC175758 180-QJ-YN-CHN-2019-CV-A4 中国 2019 LC603077 SC07-282 中国 2007 LC175762 56/QJ/YN/CHN/2020/CV-A4 中国 2020 LC626199 JL-CHN-H-CVA4-1/2007 中国 2007 JQ715710 180/QJ/YN/CHN/2020/CV-A4 中国 2020 LC626238 1-E9-CA4 中国 2008 KT353722 69-QJ-YN-CHN-2021-CVA4 中国 2021 LC707407 F1212/SD/CHN/2008 中国 2008 KY978537 189-QJ-YN-CHN-2021-CVA4 中国 2021 LC707448 JB141230145 中国 2008—2012 KC867064 23-QJ-YN-CHN-2021-CVA4 中国 2021 LC707391 CVA4/SZ/CHN/09 中国 2009 HQ728260 表 3 194R3/YN/CHN/2022分离株与其他CVA4病毒分离株不同基因组区域的同源性比较
Table 3 Homology comparison of different genomic regions between 194R3/YN/CHN/2022 and other CVA4 strains
基因区域 血清型 毒株 核苷酸同源性(%) 基因登录号 5’UTR CVA4 CV-A4/2018/herpangina/25 98.39 MN964081.1 VP4 CVA4 S270/Changsha/CHN/2019 99.52 MT920660.1 VP2 CVA4 CV-A4/2018/herpangina/16 99.48 MN964082.1 VP3 CVA4 JN20186/Shandong/China/2020 98.89 ON730850.1 VP1 CVA4 SJ2021-41/SH/CHN/CVA4/2021 97.92 MT591575.1 2A CVA4 E140/YN/CHN/2019 97.60 MT591575.1 2B CVA4 CV-A4/2018/herpangina/16 98.32 MN964082.1 2C CVA4 JN20186/Shandong/China/2020 98.48 ON730850.1 3A CVA4 JN19075/Shandong/China/2019 98.45 ON730874.1 3B CVA4 CV-A4/2018/herpangina/16 98.48 MN964082.1 3C CVA4 JN19737/Shandong/China/2019 98.18 ON730851.1 3D CVA4 JN19737/Shandong/China/2019 98.56 ON730851.1 3’UTR CVA4 CV-A4/2018/herpangina/25 98.81 MN964081.1 P1 CVA4 JN20186/Shandong/China/2020 98.28 ON730850.1 P2 CVA4 JN20186/Shandong/China/2020 97.81 ON730850.1 P3 CVA4 JN19737/Shandong/China/2019 98.41 ON730851.1 基因组 CVA4 JN20186/Shandong/China/2020 98.20 ON730850.1 -
[1] Guo W, Xu DH, Cong SR, et al. Co-infection and Enterovirus B: post EV-A71 mass vaccination scenario in China[J]. BMC Infect Dis, 2022, 22(1): 671. doi: 10.1186/s12879-022-07661-3 [2] Wieczorek M, Krzysztoszek A. Molecular characterization of Enteroviruses isolated from acute flaccid paralysis cases in Poland, 1999-2014[J]. Pol J Microbiol, 2017, 65(4): 443-450. [3] Liu YH, Chen JW, Zhang M, et al. Coxsackievirus B: the important agent of hand, foot, and mouth disease[J]. J Med Virol, 2023, 95(3): e28669. doi: 10.1002/jmv.28669 [4] Drago F, Ciccarese G, Gariazzo L, et al. Acute localized exanthem due to Coxsackievirus A4[J]. Infez Med, 2017, 25(3): 274-276. [5] Akuzawa N, Harada N, Hatori T, et al. Myocarditis, hepatitis, and pancreatitis in a patient with Coxsackievirus A4 infection: a case report[J]. Virol J, 2014, 11: 3. doi: 10.1186/1743-422X-11-3 [6] Huang J, Liao QH, Ooi MH, et al. Epidemiology of recurrent hand, foot and mouth disease, China, 2008-2015[J]. Emerg Infect Dis, 2018, 24(3): 432-442. [7] Li JS, Dong XG, Qin M, et al. Outbreak of febrile illness caused by Coxsackievirus A4 in a nursery school in Beijing, China[J]. Virol J, 2015, 12: 92. doi: 10.1186/s12985-015-0325-1 [8] 陈俊薇, 张名, 刘煜菡, 等. 云南株埃可病毒6型的全基因序列分析[J]. 中国病原生物学杂志, 2023, 18(8): 869-874. Chen JW, Zhang M, Liu YH, et al. Complete gene sequence analysis of 8 Echovirus 6 strain isolates in Yunnan Province, China[J]. Journal of Pathogen Biology, 2023, 18(8): 869-874. [9] Oberste MS, Peñaranda S, Maher K, et al. Complete genome sequences of all members of the species Human enterovirus A[J]. J Gen Virol, 2004, 85(Pt 6): 1597-1607. [10] Ji TJ, Guo Y, Lv LK, et al. Emerging recombination of the C2 sub-genotype of HFMD-associated CV-A4 is persistently and extensively circulating in China[J]. Sci Rep, 2019, 9(1): 13668. doi: 10.1038/s41598-019-49859-7 [11] Zhang M, Chen X, Wang W, et al. Genetic characteristics of Coxsackievirus A6 from children with hand, foot and mouth disease in Beijing, China, 2017-2019[J]. Infect Genet Evol, 2022, 106: 105378. doi: 10.1016/j.meegid.2022.105378 [12] 吴绮青, 何家明. EV71疫苗预防接种对手足口病的流行病学特征及临床症状的影响[J]. 医学食疗与健康, 2022, 20(7): 180-182, 193. Wu QQ, He JM. Effect of EV71 vaccination on epidemiological characteristics and clinical symptoms of hand, foot and mouth disease[J]. Medical Diet and Health, 2022, 20(7): 180-182, 193. [13] Li J, Ni N, Cui YN, et al. An outbreak of a novel recombinant Coxsackievirus A4 in a kindergarten, Shandong province, China, 2021[J]. Emerg Microbes Infect, 2022, 11(1): 2207-2210. doi: 10.1080/22221751.2022.2114855 [14] 甄若楠, 张颖, 谢华萍, 等. 2010-2012年广州市柯萨奇病毒A4、A10型VP1基因特征分析[J]. 中华预防医学杂志, 2014, 48(6): 445-450. doi: 10.3760/cma.j.issn.0253-9624.2014.06.005 Zhen RN, Zhang Y, Xie HP, et al. Sequence analysis of VP1 region of Coxsackievirus A4 and Coxsackievirus A10 in Guangzhou city, 2010-2012[J]. Chinese Journal of Preventive Medicine, 2014, 48(6): 445-450. doi: 10.3760/cma.j.issn.0253-9624.2014.06.005 [15] Hu YF, Yang F, Du J, et al. Complete genome analysis of Coxsackievirus A2, A4, A5, and A10 strains isolated from hand, foot, and mouth disease patients in China revealing frequent recombination of human enterovirus A[J]. J Clin Microbiol, 2011, 49(7): 2426-2434. doi: 10.1128/JCM.00007-11 [16] Wang M, Li J, Yao MX, et al. Genome analysis of coxsac-kievirus A4 isolates from hand, foot, and mouth disease cases in Shandong, China[J]. Front Microbiol, 2019, 10: 1001. doi: 10.3389/fmicb.2019.01001 [17] 刘艳艳, 周永明, 赵云珍, 等. 昆明市2018-2021年手足口病病例中柯萨奇病毒A组4型流行情况及VP1编码序列基因特征分析[J]. 中国病毒病杂志, 2022, 12(5): 364-370. Liu YY, Zhou YM, Zhao YZ, et al. Prevalence of Coxsac-kievirus A4 among patients with hand, foot, and mouth di-sease in Kunming in 2018-2021 and the phylogenetic analysis of its VP1 coding gene sequence[J]. Chinese Journal of Viral Diseases, 2022, 12(5): 364-370. [18] Oberste MS, Maher K, Kilpatrick DR, et al. Molecular evolution of the human Enteroviruses: correlation of serotype with VP1 sequence and application to picornavirus classification[J]. J Virol, 1999, 73(3): 1941-1948. doi: 10.1128/JVI.73.3.1941-1948.1999 [19] Feng XB, Guan WC, Guo YF, et al. Genome sequence of a novel recombinant Coxsackievirus A6 strain from Shanghai, China, 2013[J]. Genome Announc, 2015, 3(1): e01347-14. [20] Wu Y, Yeo A, Phoon MC, et al. The largest outbreak of hand; foot and mouth disease in Singapore in 2008: the role of Enterovirus 71 and Coxsackievirus A strains[J]. Int J Infect Dis, 2010, 14(12): e1076-e1081. doi: 10.1016/j.ijid.2010.07.006 [21] Yang F, Zhang T, Hu YF, et al. Survey of Enterovirus infections from hand, foot and mouth disease outbreak in China, 2009[J]. Virol J, 2011, 8: 508. doi: 10.1186/1743-422X-8-508 [22] 宫金伶, 张秀莲, 肖思颖, 等. 青岛市7株柯萨奇病毒A4型分离株全基因特征分析[J]. 中国病毒病杂志, 2022, 12(5): 376-380. Gong JL, Zhang XL, Xiao SY, et al. Whole genome sequencing of 7 strains of Coxsackievirus A4 isolated in Qingdao[J]. Chinese Journal of Viral Diseases, 2022, 12(5): 376-380. [23] 李云逸, 崔心怡, 费洁, 等. 2020-2021年上海地区分离株柯萨奇病毒A4基因型的全基因组分析[J]. 中国病原生物学杂志, 2023, 18(8): 875-880. Li YY, Cui XY, Fei J, et al. Genome analysis of recombinant Coxsackievirus A4 genotype in Shanghai from 2020 to 2021[J]. Journal of Pathogen Biology, 2023, 18(8): 875-880. [24] 王东艳, 徐艺, 张勇, 等. 陕西省急性弛缓性麻痹病例中柯萨奇病毒A组4型的基因特征[J]. 病毒学报, 2016, 32(2): 145-149. Wang DY, Xu Y, Zhang Y, et al. Genetic characteristics of Coxsackievirus group a type 4 isolated from patients with acute flaccid paralysis in Shaanxi, China[J]. Chinese Journal of Virology, 2016, 32(2): 145-149. [25] 史晓燕, 汪照国, 宫金伶, 等. 2009-2015年青岛市手足口病病原中柯萨奇病毒A4型VP1区基因特征分析[J]. 中国病毒病杂志, 2017, 7(2): 124-128. Shi XY, Wang ZG, Gong JL, et al. Genetic characteristics of the VP1 region of Coxsackievirus A4 in HFMD in Qingdao city of China from 2009 to 2015[J]. Chinese Journal of Viral Di-seases, 2017, 7(2): 124-128.

 下载:
下载: